Auch wenn ich langsam die Nase voll von Corona habe, konnte ich es als Hobby Data-Scientist natürlich trotzdem nicht lassen, die vorhandenen Schweizer COVID-19 Daten mal genauer zu untersuchen. In diesem Post zeige ich, wie leicht man mit Python, Libraries wie matplotlib und pandas und der interaktiven Jupyter Umgebung eine sehr einfache Datenanalyse durchführen kann.
📜 Daten
Der erste Schritt ist auch gleich der schwierigste: An die Daten kommen. Zum Glück hat ein Github User bereits eine CSV Datei mit der Anzahl COVID-19 erkrankten Schweizer bereitsgestellt. Danke 😃
Patientendaten wie Geschlecht, Alter, Krankheiten etc. wären natürlich sehr interessant, sind aber vertraulich und darum unzugänglich und keine Option für uns. Auch nicht-vertrauliche Daten wie Anzahl Spitalbetten, Pfleger/-innen oder Beatmungsgeräte pro Kanton sind nur sehr schwer auffindbar. Deshalb, und aus Faulheit, beschränken wir uns für den Moment auf die bereits vorhandene CSV Datei.
# covid-19 cases per date and canton
data = pd.read_csv('https://raw.githubusercontent.com/daenuprobst/covid19-cases-switzerland/master/covid19_cases_switzerland.csv')
data['Date'] = pd.to_datetime(data['Date'], dayfirst=True)
data = data.dropna()
data.head()
Hier ein Auszug der Daten:
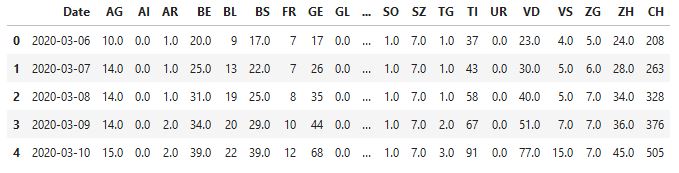
📈 Erste Auswertungen
Mit matplotlib können wir Plots erstellen, indem wir einfach einen Vektor mit Werten für die X-Achse (nachfolgend data['Date'], also die Datumsspalte), und einen Vektor für die Werte der Y-Achse angeben (nachfolgend data['CH'], das ist die Total-Spalte für die ganze Schweiz). Schauen wir zum Beispiel mal, wie sich die Anzahl COVID-19 Fälle in der Schweiz entwickelt:
plt.title('Total infizierte Personen in der Schweiz')
plt.plot(data['Date'], data['CH'])
plt.xticks(rotation=45)
plt.show()
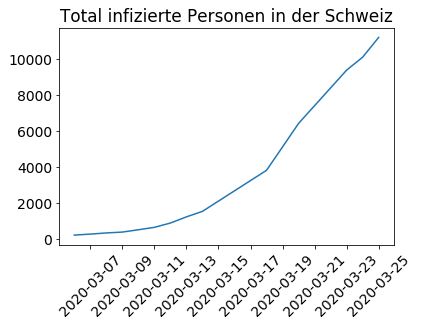
Die Kurve verläuft exponentiell - war auch nicht anders zu erwarten, ist ja eine Pandemie 😵🌍.
Anstatt nur einem Vektor können wir dem Plot auch mehrere Vektoren (bzw. eine Matrix) als Werte für die Y-Achse mitgeben. Nachfolgend geben wir alle Spalten von AG bis ZH mit, also die Daten aller Kantone, jedoch nicht die Total-Spalte CH. So erhalten wir pro Kanton eine Kurve.
plt.figure(figsize=(10,10))
plt.title('Infizierte Personen pro Kanton')
ys = data.loc[:, 'AG':'ZH']
plt.plot(data['Date'], ys)
plt.xticks(rotation=45)
plt.legend(ys.columns, fontsize=12)
plt.show()
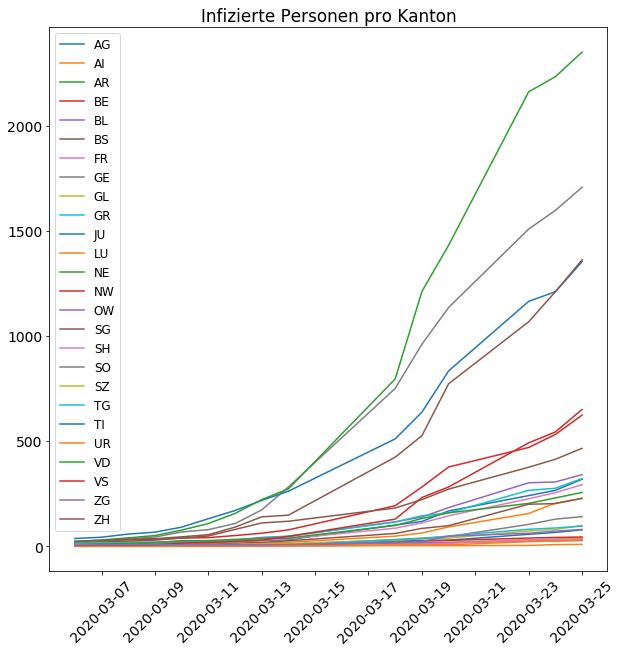
🗃️ Hinzufügen von Stammdaten
In den vorherigen Plots sehen wir die absolute Anzahl an COVID-19 Fällen - langweilig, das können wir auch in den News nachlesen. Interessanter wäre zum Beispiel der Anteil infizierter Personen pro Kanton. Dazu brauchen wir aber zuerst noch einige Stammdaten der Kantone. Wikipedia zu Hülf! Ich hab die Fläche, Population, Dichte sowie das BIP (because why not) von hier herauskopiert und in ein Dataframe cantons gepackt. Nachfolgend ein Ausschnitt davon:

Jetzt können wir diese Daten mit den Corona Daten verknüpfen und zum Beispiel den infizierten Bevölkerungsanteil pro Tag ausrechnen. Wir können ganz einfach den / operator verwenden, um die Anzahl Fälle durch die Population zu teilen. Funktioniert in Python auch für Dataframes, solange die Grösse der Vektoren/Matrizen übereinstimmt.
# percentage of infected population per canton
plt.figure(figsize=(10,10))
plt.title('Infizierter %-Anteil der Kantonspopulation')
ys = data.loc[:, 'AG':'ZH'] / cantons.iloc[:-1,:]['population'].values*100 #iloc to remove the CH column
plt.plot(data['Date'], ys)
plt.legend(ys.columns, fontsize=12)
plt.xticks(rotation=45)
plt.show()
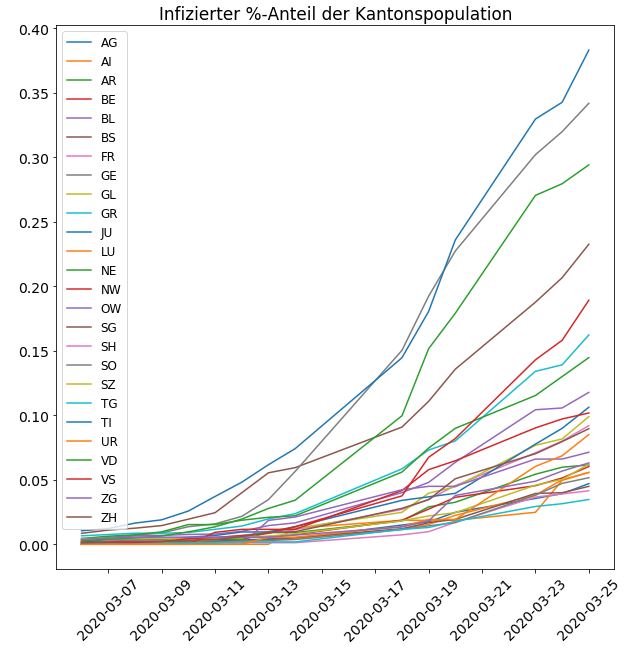
Das Bild sieht ähnlich aus wie vorhin, nur die Verteilung der Kantone ist ein wenig anders.
📊 Beschränkung auf aktuellste Werte
So weit so gut. Vergessen wir mal die Vergangenheit und konzentrieren uns nur auf die aktuellsten Daten und berechnen den infizierten Bevölkerungsanteil sowie die Anzahl infizierten Personen pro Quadratkilometer pro Kanton. Auch hierzu verwenden wir einfach die üblichen mathematischen Operatoren / und *.
# update cantons with most recent information
cantons['infected'] = data.iloc[-1, 1:].values.astype(int)
cantons['infected_percentage'] = cantons['infected'] / cantons['population'].values*100
cantons['infected_per_km2'] = cantons['infected'] / cantons['area'].values
cantons.head()
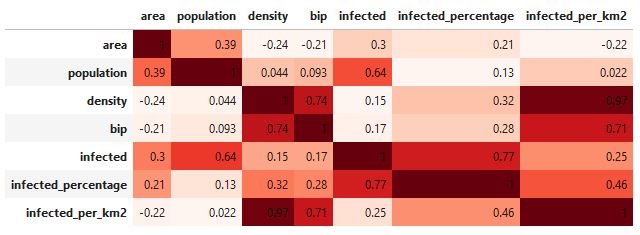
Nur der Vollständigkeit halber zeige ich nachfolgend auch, wie der Pearson Korrelationskoeffizient berechnet und visualisiert werden kann. In diesem Beispiel ist jedoch keine interessante Korrelation vorhanden.
corr = cantons.corr()
corr.style.background_gradient(cmap='Reds').set_precision(2)
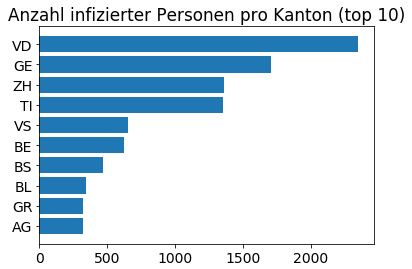
🔝 And the winner is …
Wir wissen bereits, welche Kantone die meisten infizierten Personen haben, Waadt führt zurzeit:
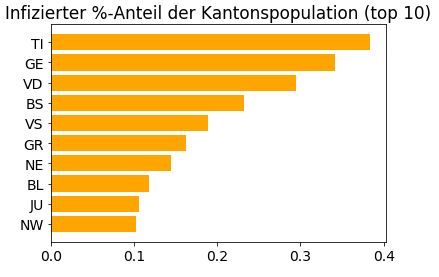
Aber hier noch mal ein bisschen übersichtlicher, welche Kantone den grössten Anteil an Infizierten haben. Hier rutscht der Kanton Tessin gleich an die Spitze.
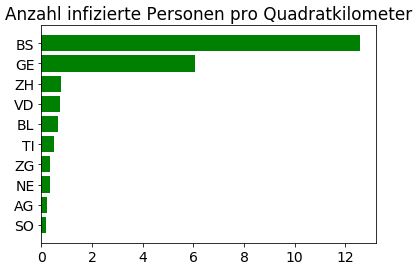
🏠 Home sweet home
Wir werden wahrscheinlich bald überfüllte Spitäler haben… Darum ist es wahrscheinlich keine schlechte Idee, möglichst zu Hause zu bleiben für den Moment. Wenn man nur die Fläche der Kantone beachtet, dann gilt das vor allem für unsere kleinen aber dicht besiedelten Kantone Basel-Stadt und Genf!
plt.title('Anzahl infizierte Personen pro Quadratkilometer')
ys = cantons.sort_values('infected_per_km2').tail(10)
plt.barh(ys['name'], ys['infected_per_km2'], color='green')
plt.show()
Danke fürs Lesen 😁✌🏻
PS: Wenn ich mal dazu komme, werde ich mich noch einer kleinen Domino-Effekt-Simulation widmen um das Exponentielle Wachstum einer solchen Pandemie möglichst verständlich zu visualisieren… Oder falls das bereits jemand macht oder gemacht hat, let me know! Nachtrag: here you go ;)
